MetaMapの使い方: 医療文書からUMLS conceptを抽出するには
1. 目的
ここで取り上げるのは医学論文, 診療記録, 退院サマリーなどの医療文書に対して固有表現抽出を行うMetaMapという医療言語処理ツールです.
このツールを使うと任意の医療文書から疾患名, 薬剤名, 治療名などを抽出することができます.
一見, ただ目的の用語を抽出するだけなら部分一致検索でもよいと思えるかもしれません.
しかし, 医療文書には
- 表記ゆれが存在する (corona virus, corona-virus, coronavirusなど)
- ほとんど同じ実体を表す用語が複数存在する (pneumonia, lung inflammation, pulmonary inflammationなど)
という特徴があり, そのすべてを想定して対処するのは容易ではありません.
ありがたいことに, 米国国立衛生研究所 National Institutes of Health (NIH) が提供しているMetaMapというツールには, これらの表記の差異を吸収する (=正規化する) 機能が備わっています.
MetaMapの使い方に言及している資料があまり無かったので, 備忘録も兼ねて簡単な記事にしました.
2. Unified Medical Language System (UMLS)
正確にはMetaMapが検出するのは用語というよりはUMLSに収載されているconceptです.
UMLSはNIHが提供するメタシソーラスで, ICD-10, SNOMED-CT, MeSHなどの異なるシソーラス同士を連結することによって言語資源どうしの壁を超えた横断的な検索が可能となっています.
同じ実体は Contept Unique Identifier (CUI) というコードによって紐づけられています.
3. MetaMapの実行(1): Webブラウザから
まず最初に UMLS License を取得しておく必要があります.
こちらのページにアクセスし, 右上の Sign Up から登録を済ませてください.
登録が無事に済んだら, WebブラウザでMetaMapを実行してみましょう.
以下ではこちらの論文のIntroductionの最初の2文をサンプルとして使用していきます.
In December 2019, an outbreak caused by a new coronavirus was started in Wuhan, Hubei province of China that led to a pandemic emergence according to the World Health Organization (WHO) on March 11, 2020. According to the phylogenetic studies, the pathogen was named severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) and the disease was called coronavirus disease 2019 (COVID-19). Reports have shown different signs among the patients with COVID-19 among which fever and cough were most common.
3-1. Interactive MetaMap
まず interactive MetaMap にアクセスし, 右の入力欄に固有表現抽出を行いたいテキストを入力しましょう.

画面が切り替わり, 結果が Results 欄に出力されます.
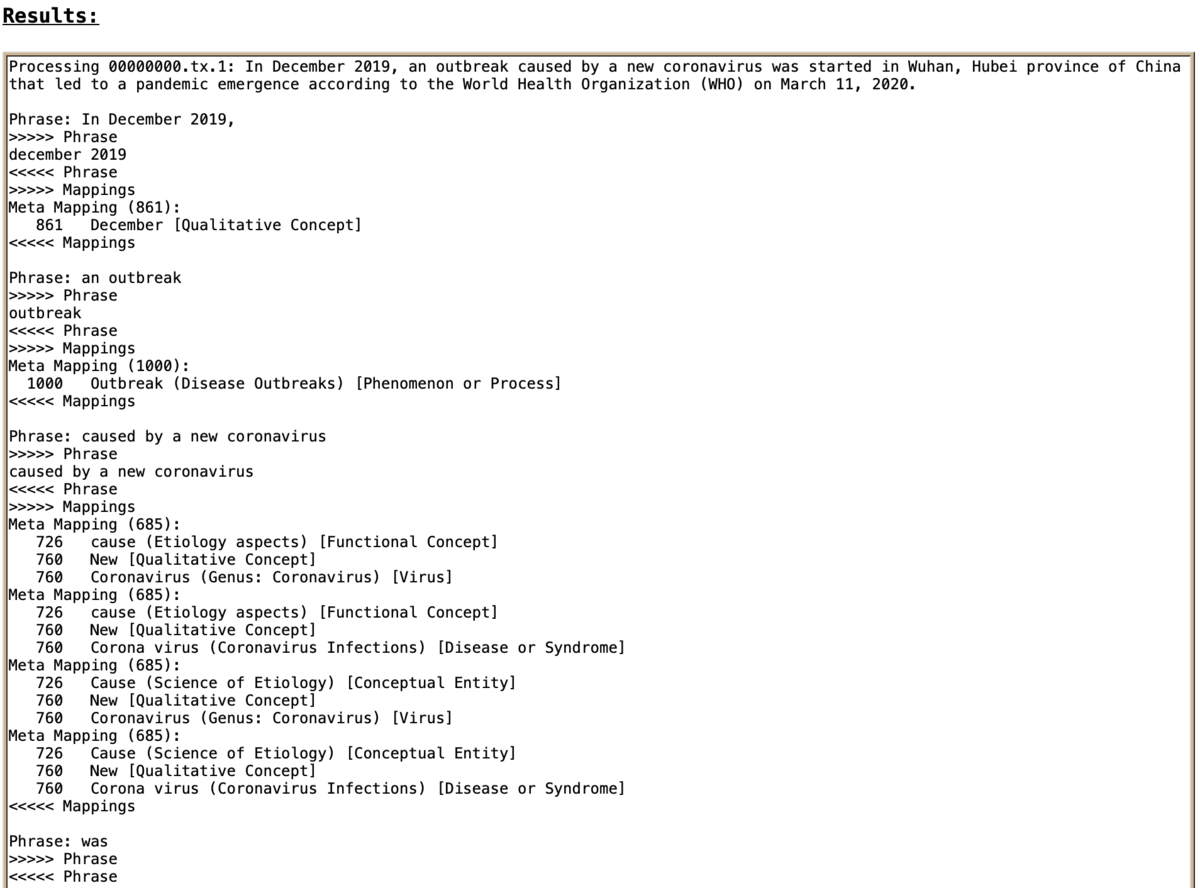
出力結果はおおむね句ごとに区切られて以下のように表示されています.
Phrase: もとのテキストの句 >>>>> Phrase UMLS conceptが検出された箇所 <<<<< Phrase >>>>> Mappings UMLS conceptの検出結果の候補1 (Mapping score): Mapping score 正規化後の表現 [Semantic Type] Mapping score 正規化後の表現 [Semantic Type] ... UMLS conceptの検出結果の候補2 (Mapping score): ... <<<<< Mappings
3-2. 表示オプション(1)
以下の欄から出力結果の表示形式を変更することもできます.
Show CUIs (-I)- デフォルトの表示形式から装飾 (">>>>>", "<<<<<") を省略する.
Display Tagger Output (-T)Show CUIsに加えて品詞タグ付け情報を表示(1行目に文, 2行目に品詞タグ).
Formal Tagger Output (-F)Show CUIsに加えて品詞タグ付け情報を表示(配列形式).
Syntax (-x)Show CUIsに加えて品詞タグ付け+表出形+正規化表現+分かち書きを表示.
Display Variants (-v)Show CUIsに加えて表記ゆれ情報を表示.
Hide Plain Syntax (-p)Show CUIsからPhraseを省略してMeta mappingだけを表示.
Show Candidates (-c)Show CUIsに加えてCandidatesを表示.
Number Candidates (-n)- (未検証)
Number Mappings (-f)Show CUIsに加えてMeta mappingに連番を振る.
Short Semantic Types (-s)Show CUIsのsemantic typeを略語で表示する.
Show Concept's Sources (-G)Show CUIsの各UMLS conceptに対してそれを含むシソーラス名を表示する.
Show Acronym/Abbreviations (-j)Show CUIsの出力の冒頭に検出された略語一覧を付加する.
3-3. 表示オプション(2): Fielded MetaMap Indexing (MMI) Output (-N)
以下のオプションでより詳細な出力結果を得ることも可能です.


- 1列目: PMID.
- 2列目: "MM"という文字列.
- 3列目: MetaMap Indexing (MMI) score (max 1000.00)
- 4列目: UMLS Concept Preferred Name
- 5列目: UMLS Contept Unique Identifier (CUI)
- 6列目: Semantic Type List
- 7列目: Location: TI, AB, or TI;AB
- 8列目: Positional Information: xxx:yyy (position:length)
- 9列目: Treecode
3-4. 表示オプション(3): Machine Output (-q)
以下のような構造をもった MetaMap Machine Output (MMO) 形式での出力.
後述しますが, NIHから提供されているMetaMap実行済みMEDLINE論文データもMMO形式です.
args(コマンド)
aas(略語一覧)
neg_list
utterance('文')
phrase('句',[品詞1(正規化表現,表出形,品詞,分かち書き), 品詞2(正規化表現,表出形,品詞,分かち書き), ...])
candidates
mappings
phrase
candidates
mappings
3-5. 表示オプション(4): XMLで表示
XMLで出力することもできます. 深層学習などに利用するタグ付きコーパスの作成にも便利かと思います.
Formatted XML Output (--XMLf)- 以下のような構造 (一部を抜粋) のXML文書を出力.
Unformatted XML Output (--XMLn)- インデントや改行で整形されていないXML文書を出力.
- 文: <Uttrance> ~ </Uttrance> - 句: <Phrase> ~ </Phrase> - 品詞タグ付け情報: <SyntaxUnits> ~ </SyntaxUnits> - 品詞: <SyntaxType> ~ </SyntaxType> - 正規化表現: <LexMatch> ~ </LecMatch> - 表出形: <InputMatch> ~ </InputMatch> - 品詞: <LexCat> ~ </LexCat> - UMLS concept情報: <Mappings> ~ </Mappings> - score: <CandidateScore> ~ </CandidateScore> - CUI: <CandidateCUI> ~ </CandidateCUI> - 表出形: <MatchedWord> ~ </MatchedWord> - semantic type: <SemType> ~ </SemType> - シソーラス情報: <Sources> ~ </Sources> - シソーラス名: <Source> ~ </Source>
3-6. 表示オプション(5): JSONで表示
JSONでの出力も可能です.
Formatted JSON Output (--JSONf 2)- 以下のような構造 (一部を抜粋) のJSONを出力.
Unformatted JSON Output (--JSONn)- インデントや改行で整形されていないJSONを出力.
"Utterances" : [ { //文 "Phrases" : [ { //句 "Phrasetext" : hogehoge, "SyntaxUnits" : [ { //品詞タグ "SyntaxType" : 品詞, "LexMatch" : 正規化表現 "InputMatch" : 表出形 "LexCat" : 品詞 }, ... ], "Mappings" : [ { //UMLS concept情報 "MappintScore" : score, "MappintCandidates" : { "CandidateCUI" : CUI, "CandidateMatched" : 表出形, "SemTypes" : [semantic type], "Sources" : [収載シソーラス] }, ... ], }, ... ]
3-7. 固有表現抽出のオプション: シソーラスの指定/除外 Restrict to Sources (-R) / Exclude Sources (-e)
デフォルトではUMLSに収載されているあらゆる項目が抽出されますが,
「ICD-10の疾患名だけ」など, 特定のシソーラスの収載項目だけを指定した固有表現抽出も可能です.
3-8. 固有表現抽出のオプション: Semantic Typesの指定/除外 Restrict to Semantic Type(s) (-J) / Exclude Semantic Type(s) (-k)
UMLSの各項目には「疾患名」「解剖学的名称」「薬剤名」などのいずれであるかを表す Semantic Type が付与されています.
www.nlm.nih.gov
これを利用して, 特定の種類の固有表現だけを抽出することも可能です.
- 例1:薬剤名だけ抽出したいとき
- 以下のSemantic Typesだけを含めるようにする:
- Antibiotics (
antb) - Organic Chemical (
orch) - Pharmacologic Substance (
phsu)
- Antibiotics (
- 以下のSemantic Typesだけを含めるようにする:
4. MetaMapの実行(2): コマンドラインで実行する
MetaMapをローカルで実行することもできます.
固有表現抽出を行いたい医療文書が大量にある場合や, コマンドラインでの操作に慣れている場合にお勧めです.
- 実行環境:
- Mac OS X 10.14.6
- Terminal 2.9.5
https://metamap.nlm.nih.gov/MainDownload.shtmlにアクセスし, まずMetaMapの本体をダウンロードしてきましょう.
サイズが大きいので注意してください (tarアーカイブは約1.6GB, 展開後のサイズは約7GBあります).
ダウンロード後は以下のような手順でMetaMapを実行することができます.
# アーカイブを展開 $ tar xf public_mm_darwin_main_2018.tar.bz2 # インストール $ cd public_mm $ ./bin/install.sh # Javaが未インストールならJavaもインストール (以下はHomebrewの場合) $ brew cask install java # SKR/Medpost Part-of-Speech Tagger Serverを起動 $ ./bin/skrmedpostctl start >>> Starting skrmedpostctl: started. # "lung cancer" という文に対して固有表現抽出 $ echo "lung cancer" | ./bin/metamap >>> Phrase: lung cancer Meta Mapping (1000): 1000 C0242379:Lung Cancer (Malignant neoplasm of lung) [Neoplastic Process] Meta Mapping (1000): 1000 C0684249:LUNG CANCER (Carcinoma of lung) [Neoplastic Process] Meta Mapping (1000): 1000 C1306460:Lung cancer (Primary malignant neoplasm of lung) [Neoplastic Process] # 先ほど挙げた各種オプションも適用可能 $ echo "lung cancer" | ./bin/metamap -I -T -p -f -G -s >>> lung cancer noun noun 1. Meta Mapping (1000): 1000 C0242379:Lung Cancer (Malignant neoplasm of lung {CHV,COSTAR,LCH_NW,MEDLINEPLUS,MSH,MTH,NCI,NCI_CTRP,NLMSubSyn,OMIM,SNMI,SNOMEDCT_US}) [neop] 2. Meta Mapping (1000): 1000 C0684249:LUNG CANCER (Carcinoma of lung {AOD,CHV,COSTAR,CSP,CST,HPO,ICD10CM,LNC,MTH,NCI,NCI_CTEP-SDC,NCI_CTRP,NCI_NCI-GLOSS,NLMSubSyn,OMIM,PDQ,SNOMEDCT_US}) [neop] 3. Meta Mapping (1000): 1000 C1306460:Lung cancer (Primary malignant neoplasm of lung {MTH,NLMSubSyn,SNOMEDCT_US}) [neop] # 使い終わったらSKR/Medpost Part-of-Speech Tagger Serverをシャットダウン $ ./bin/skrmedpostctl stop >>> Stopping skrmedpostctl: Stopping Tagger Server process.. Process xxxxx stopped # SKR/Medpost Part-of-Speech Tagger Serverのシャットダウン忘れを確認 # /usr/bin/java ... taggerServer が返るときはシャットダウンできていない $ ps -ef | grep java
5. MetaMapの結果だけが欲しいときは?
MEDLINE論文に対してMetaMapを実行したい場合は, 出力結果が MetaMapped MEDLINE Baselines で提供されているため, そちらを利用するのも手です.
こちらを使うと自力で「MEDLINE論文を取得 -> 本文を抽出 -> MetaMapを実行」を行う手間が省けます.
なお, 出力結果はMachine Output形式で提供されています (上記3-4.を参照).