Dockerことはじめ: 初心者がコンテナを作ったり壊したりして覚えてみる
0. はじめに
0-1. Dockerを使おうとしたきっかけ
- 私は普段はPyTorch使いなのですが, 研究の都合上, どうしてもTensorFlowを使わないとならない場面に遭遇しました.
- しかし, とある事情で, NAISTの共用GPUサーバーの環境ではTensorFlowが使えないことが発覚してしまいました.
- つまり, TensorFlowはどうしても使いたい, しかし, かといって共用サーバーの環境を個人の都合で勝手に弄りたくない, というジレンマに陥ったわけです.
- そこで, TensorFlowが公式に提供しているDocker Imageを使ってみることにしました.
0-2. この記事で書いていること/書いていないこと
- 書いていること
- Dockerのごくごく初歩 (Docker HubからのImageの入手とコンテナの作成)
- 書いていないこと
- コンテナの概念
- Dockerのインストール方法
0-3. 動作環境
1. Docker Imageを入手したり捨てたりしてみる
1-0. 準備
仮に, 研究プロジェクトの各種ファイルやスクリプトを/home/me/myproject以下に保存しているものとします.
me@server ~ $ cd myproject me@server myproject $ docker --version # Dockerのバージョン確認 Docker version 19.03.8, build afacb8b
1-1. Docker Imageの入手
TensorFlow最新版のDocker Imageで, GPUとJupyterに対応したものをダウンロードしてきます.
# Docker Imageの名称は <リポジトリ名>:<タグ名> のようになっている me@server myproject $ docker pull tensorflow/tensorflow:latest-gpu-jupyter # ダウンロードしたDocker Imageの一覧 me@server myproject $ sudo docker images REPOSITORY TAG IMAGE ID CREATED SIZE tensorflow/tensorflow latest-gpu-jupyter 0123456789ab 8 weeks ago 3.99GB
1-2. Docker Imageの削除
一応, 削除のしかたも覚えておきます.
# Docker imageを一度削除してみる me@server myproject $ sudo docker rmi 0123456789ab # もう一度同じDocker imageを取得 me@server myproject $ docker pull tensorflow/tensorflow:latest-gpu-jupyter
2. Docker コンテナを作ったり壊したりしてみる
2-1. 作成と起動(1): docker create -> docker start してみる
ものすごく雑な理解では, Docker Imageは設計図でDocker コンテナが仮想マシン本体のようなものだと思っています.
docker createでコンテナを作成してみます.
# Docker imageをもとにコンテナを作成する # --nameでコンテナ名をつける # -it オプションを渡しておくとインタラクティブなコンテナを作成できる me@server myproject $ sudo docker create -it --name tf_container tensorflow/tensorflow:latest-gpu-jupyter
docker ps -aでコンテナの一覧を確認できます.
me@server myproject $ sudo docker ps -a # STATUSが "Created" になっている (作成されただけで起動はしていない) CONTAINER ID IMAGE COMMAND CREATED STATUS PORTS NAMES 456789abcdef tensorflow/tensorflow:latest-gpu-jupyter "bash -c 'source /et…" 30 seconds ago Created tf_container
docker startでコンテナを起動します.
TensorFlowのJupyter対応版Dockerイメージでは, デフォルトではコンテナの起動と同時にJupyter Notebookが起動するようになっているようです.
# -i オプションでインタラクティブなセッションを開始できる # Jupyter Notebookがただちに立ち上がる me@server myproject $ sudo docker start -i tf_container jupyter_http_over_ws extension initialized. Listening on /http_over_websocket [I 14:41:29.417 NotebookApp] Serving notebooks from local directory: /tf [I 14:41:29.417 NotebookApp] The Jupyter Notebook is running at: [I 14:41:29.417 NotebookApp] http://456789abcdef:8888/?token=xxxxxxxxxxxxxx [I 14:41:29.417 NotebookApp] or http://127.0.0.1:8888/?token=xxxxxxxxxxxxxx [I 14:41:29.417 NotebookApp] Use Control-C to stop this server and shut down all kernels (twice to skip confirmation). [C 14:41:29.421 NotebookApp] To access the notebook, open this file in a browser: file:///root/.local/share/jupyter/runtime/nbserver-1-open.html Or copy and paste one of these URLs: http://456789abcdef:8888/?token=xxxxxxxxxxxxxx or http://127.0.0.1:8888/?token=xxxxxxxxxxxxxx
ここでは何もせず, Control+c->yでJupyter Notebookを終了してみます. するとコンテナのインタラクティブセッションも終了し, ホスト側に戻ります.
me@server myproject $
docker ps -aでもう一度コンテナの一覧をみるとコンテナの停止を確認できます.
me@server myproject $ sudo docker ps -a # STATUSが "Exited" になっている (コンテナが停止している) CONTAINER ID IMAGE COMMAND CREATED STATUS PORTS NAMES 456789abcdef tensorflow/tensorflow:latest-gpu-jupyter "bash -c 'source /et…" 30 seconds ago Exited (0) 5 seconds ago tf_container
2-2. 起動時のコマンドを変えてみる(1)
先ほどはコンテナを起動した瞬間にJupyter Notebookが立ち上がりました.
しかし, これではJupyter Notebook以外に何もできないので, かわりにコンテナ起動時にシェルが立ち上がるようにしたいです.
再びdocker startでコンテナを起動してみます.
# ここでは -i オプションを渡さずに起動する # すると, コンテナは起動するがインタラクティブセッションには移らない me@server myproject $ sudo docker start tf_container tf_container
docker exec コンテナ名 コマンドでコマンドを実行できます.
# -it オプションを渡すとインタラクティブセッションが始まる me@server myproject $ sudo docker exec -it tf_container bash
コンテナ内でシェルが立ち上がりました!
________ _______________ ___ __/__________________________________ ____/__ /________ __ __ / _ _ \_ __ \_ ___/ __ \_ ___/_ /_ __ /_ __ \_ | /| / / _ / / __/ / / /(__ )/ /_/ / / _ __/ _ / / /_/ /_ |/ |/ / /_/ \___//_/ /_//____/ \____//_/ /_/ /_/ \____/____/|__/ WARNING: You are running this container as root, which can cause new files in mounted volumes to be created as the root user on your host machine. To avoid this, run the container by specifying your user's userid: $ docker run -u $(id -u):$(id -g) args... root@456789abcdef:/tf#
ここでは何もせず, コンテナのインタラクティブセッションから抜けます.
root@456789abcdef:/tf# exit exit me@server myproject $
docker ps -aでもう一度コンテナの一覧を確認してみます.
me@server myproject $ sudo docker ps -a # STATUSは "Up" になっている (コンテナは起動したまま) CONTAINER ID IMAGE COMMAND CREATED STATUS PORTS NAMES 456789abcdef tensorflow/tensorflow:latest-gpu-jupyter "bash -c 'source /et…" 30 seconds ago Up 15 seconds 8888/tcp tf_container
2-3. 停止と削除
docker stopでコンテナを停止 -> docker rmでコンテナを削除できます.
(もしくは, docker rm -fで起動中のコンテナも強制的に削除可能)
me@server myproject $ sudo docker stop tf_container tf_container me@server myproject $ sudo docker rm tf_container tf_container
2-4. 作成と起動(3): docker run してみる
docker run イメージ名:タグ名 コマンドでコンテナの作成と起動を同時に行うことができます.
# 先ほどとはコンテナ名を変えて, tf_container_shell という名前で作成する # 末尾に起動直後に実行させたいコマンド (ここでは bash) を付加する me@server myproject $ sudo docker run -it --name tf_container_shell tensorflow/tensorflow:latest-gpu-jupyter bash
ここでは docker runに-it オプションと bash コマンドを付加したので, tf_container_shell コンテナが作成されると同時にコンテナ内でシェルが立ち上がり, そのインタラクティブセッションに移行することができます.
________ _______________ ___ __/__________________________________ ____/__ /________ __ __ / _ _ \_ __ \_ ___/ __ \_ ___/_ /_ __ /_ __ \_ | /| / / _ / / __/ / / /(__ )/ /_/ / / _ __/ _ / / /_/ /_ |/ |/ / /_/ \___//_/ /_//____/ \____//_/ /_/ /_/ \____/____/|__/ WARNING: You are running this container as root, which can cause new files in mounted volumes to be created as the root user on your host machine. To avoid this, run the container by specifying your user's userid: $ docker run -u $(id -u):$(id -g) args... root@23456789abcd:/tf#
ここでは何もせず, インタラクティブセッションから抜け, コンテナも削除します.
root@23456789abcd:/tf# exit exit me@server myproject $ sudo docker rm tf_container_shell tf_container_shell
3. おわりに
Dockerコンテナの作成, 削除, 起動, 停止についてだいぶ慣れてきました.
次はDockerコンテナ内で研究プロジェクトを動かす準備をしていきたいと思います.
次の記事はこちら
参考にした記事
https://qiita.com/tifa2chan/items/e9aa408244687a63a0ae
奈良先端大に国内留学して2ヶ月経ちました 〜生活環境の備忘録〜
いま私は医学博士課程2年目で,社会人大学院生のような立場なのですが,4月から半年間仕事をストップして,NAIST荒牧研に特別研究学生として医療言語処理を学びに国内留学しています.
大まかな理由は以下のとおりです.
- いまの環境が相当厳しい
- 自然言語処理に詳しい人が周囲におらず,相談相手がいない
- 論文を読んでつまずいた日も,実装につまずいた日も,ひたすら独学
- 病院で週5日働いており時間的制約が大きい
- 仕事を終えて研究室に直行して深夜1時すぎに帰る生活,お正月も毎日研究室に行っていた
- 自然言語処理に詳しい人が周囲におらず,相談相手がいない
- しかし,研究成果をあげるチャンスは今しかない
- まだ医療言語処理が流行り始めたばかりのタイミングなので,今ならギリギリ開拓者になれる
- おそらく3年後にはもう red ocean になっているはず
- 今のゆっくりした成長速度では先行者として走りつづけることができない
半年間収入はゼロになるし,専門医取得などのキャリアも一年遅れることになりますが,それでも構わないという強い気持ちで医局にお願いをして国内留学が実現しました.
はやくも2ヶ月経ち,とてつもなく恵まれた環境にいることを実感しています.

日本国内でもっと医療言語処理はどんどん盛り上がって欲しいですし,できれば情報系の人々だけではなく,医療関係者側からも興味をもって参入してきてほしいと願っています.
そのための一つの参考になればと思い,開始前にはわからなかった留学生活などの細かな点について備忘録を残しておきたいと思います.
研究環境
生活環境
初めて研究室見学にきた当時は,NAISTの周りに何もないのをみて「ここで生活していけるのか・・・?」と若干不安に思いましたが,案外慣れてしまえば快適です。
住環境
- ゲストハウスせんたんに宿泊しています
- 90日間まで滞在可能, 料金は90日間で9万円 (破格の安さ)
- 基本的に延長はできない
- 90日を超える場合はサイエンスプラザに宿泊場所を移すか, NAISTが借り上げている中登美団地に住む必要があります
- NAIST構内にあるので研究室に徒歩1分くらいでアクセスできる
- 週1回掃除に入ってもらえるので基本的に自分で家事をしなくてよい
- 清掃日前日になるとドアに案内と「掃除してください/起こさないでください」のタグが貼られる
- 清掃日の13時までに「掃除してください」にすればOK
- 家具は一通りそろっている
- デスク, 卓上ライト, テーブル, 椅子, 一人掛けソファ, ベッド, テレビ, DVDプレイヤー
- 冷蔵庫, 電子レンジ, やかんも部屋にある
- ポットはないのでT-FALを持って行ったほうがよい
- まな板, 洗剤, スポンジ, マグカップ, トレイは部屋にある
- 箸も1階自販機横に大量に置いてある割り箸を使えばOK
- 他の調理器具, 皿, フォーク, スプーンはない
- ラップ, アルミホイルはない
- ただし, 管理人室に頼めば一部借りられるものもあるとのこと
- バスとトイレは部屋にある
- ユニットバス
- シャンプー, ボディソープ, タオルは清掃日に補充・交換してもらえる
- 洗濯機と乾燥機は共用
- 洗濯機は3台, 乾燥機は1台
- 乾燥機は休日は混み合う. 平日のほうが使いやすい
- 洗濯ネット, 洗濯用洗剤, 柔軟剤は自分で買う必要がある
- 有線LANケーブルが壁から生えている
- 学内無線LANが届くので基本的にお世話になる機会はない
- 1階にジムが併設されている
- しかしCOVID-19の影響で閉鎖中. 当分再開の見込みはなさそう
食事事情
平日 (私の場合)
- 朝食: 構内のデイリーヤマザキで前日に買っておくとよい
- もしくは朝8時の開店を待って買う
- デイリーヤマザキのサラダサーモンはいいぞ
- 昼食: 学食
- 日替わり定食は11:30販売開始. 数に限りがある
- 日替わり定食が売り切れていた場合は通常メニューから選ぶことになる
- しかし, 通常メニューはバリエーションが少ないので注意
- 日替わり定食を逃し続けると週に5回くらい親子丼を食べることになる
- 夕食: 学食
- 日替わり定食は17:30販売開始. 数に限りがある
- 日替わり定食が売り切れていた場合は通常メニューから選ぶことになる
- しかし, 通常メニューはバリエーションが少ないので注意
- 日替わり定食を逃し続けると(以下略)
- ラストオーダー20:30, 閉店21:00
- デイリーヤマザキも21:00まで開いてはいるが, 食べ物はあまり残っていないことが多い
- 朝食: 構内のデイリーヤマザキで前日に買っておくとよい
土休日 (私の場合)
自炊
- 私自身は土休日に中食をするくらいであまり自炊していないが, やろうと思えばできそう
- 調理器具については↑, 食料品については↓を参照
外食
- NAIST徒歩圏内には外食できる店はほぼない
出前
買い物, 理美容, 移動
以下は公共交通機関 or 徒歩での移動を想定
- もっと自由に移動したい場合は車, 原付, 電動自転車などを推奨. ただし無くても生活はできる
- アップダウンが激しいので電動なしの自転車はおすすめしない
- ちなみにカーシェアのステーションはふもとの団地側にはあるがNAIST周辺にはない
食料品など
家電量販店
- 近鉄奈良周辺か大阪方面に行かないと大きな量販店はない (やや大変)
書店
その他の買い物
美容室
- 南生駒駅徒歩数分のヘアメイク FEEL 本店がかなりオススメです
- 受賞歴のあるスタイリストさんがいる. マッサージが気持ち良い
- 南生駒駅徒歩数分のヘアメイク FEEL 本店がかなりオススメです
駅までの移動まとめ
郵便物と通販
MetaMapの使い方: 医療文書からUMLS conceptを抽出するには
1. 目的
ここで取り上げるのは医学論文, 診療記録, 退院サマリーなどの医療文書に対して固有表現抽出を行うMetaMapという医療言語処理ツールです.
このツールを使うと任意の医療文書から疾患名, 薬剤名, 治療名などを抽出することができます.
一見, ただ目的の用語を抽出するだけなら部分一致検索でもよいと思えるかもしれません.
しかし, 医療文書には
- 表記ゆれが存在する (corona virus, corona-virus, coronavirusなど)
- ほとんど同じ実体を表す用語が複数存在する (pneumonia, lung inflammation, pulmonary inflammationなど)
という特徴があり, そのすべてを想定して対処するのは容易ではありません.
ありがたいことに, 米国国立衛生研究所 National Institutes of Health (NIH) が提供しているMetaMapというツールには, これらの表記の差異を吸収する (=正規化する) 機能が備わっています.
MetaMapの使い方に言及している資料があまり無かったので, 備忘録も兼ねて簡単な記事にしました.
2. Unified Medical Language System (UMLS)
正確にはMetaMapが検出するのは用語というよりはUMLSに収載されているconceptです.
UMLSはNIHが提供するメタシソーラスで, ICD-10, SNOMED-CT, MeSHなどの異なるシソーラス同士を連結することによって言語資源どうしの壁を超えた横断的な検索が可能となっています.
同じ実体は Contept Unique Identifier (CUI) というコードによって紐づけられています.
3. MetaMapの実行(1): Webブラウザから
まず最初に UMLS License を取得しておく必要があります.
こちらのページにアクセスし, 右上の Sign Up から登録を済ませてください.
登録が無事に済んだら, WebブラウザでMetaMapを実行してみましょう.
以下ではこちらの論文のIntroductionの最初の2文をサンプルとして使用していきます.
In December 2019, an outbreak caused by a new coronavirus was started in Wuhan, Hubei province of China that led to a pandemic emergence according to the World Health Organization (WHO) on March 11, 2020. According to the phylogenetic studies, the pathogen was named severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) and the disease was called coronavirus disease 2019 (COVID-19). Reports have shown different signs among the patients with COVID-19 among which fever and cough were most common.
3-1. Interactive MetaMap
まず interactive MetaMap にアクセスし, 右の入力欄に固有表現抽出を行いたいテキストを入力しましょう.

画面が切り替わり, 結果が Results 欄に出力されます.
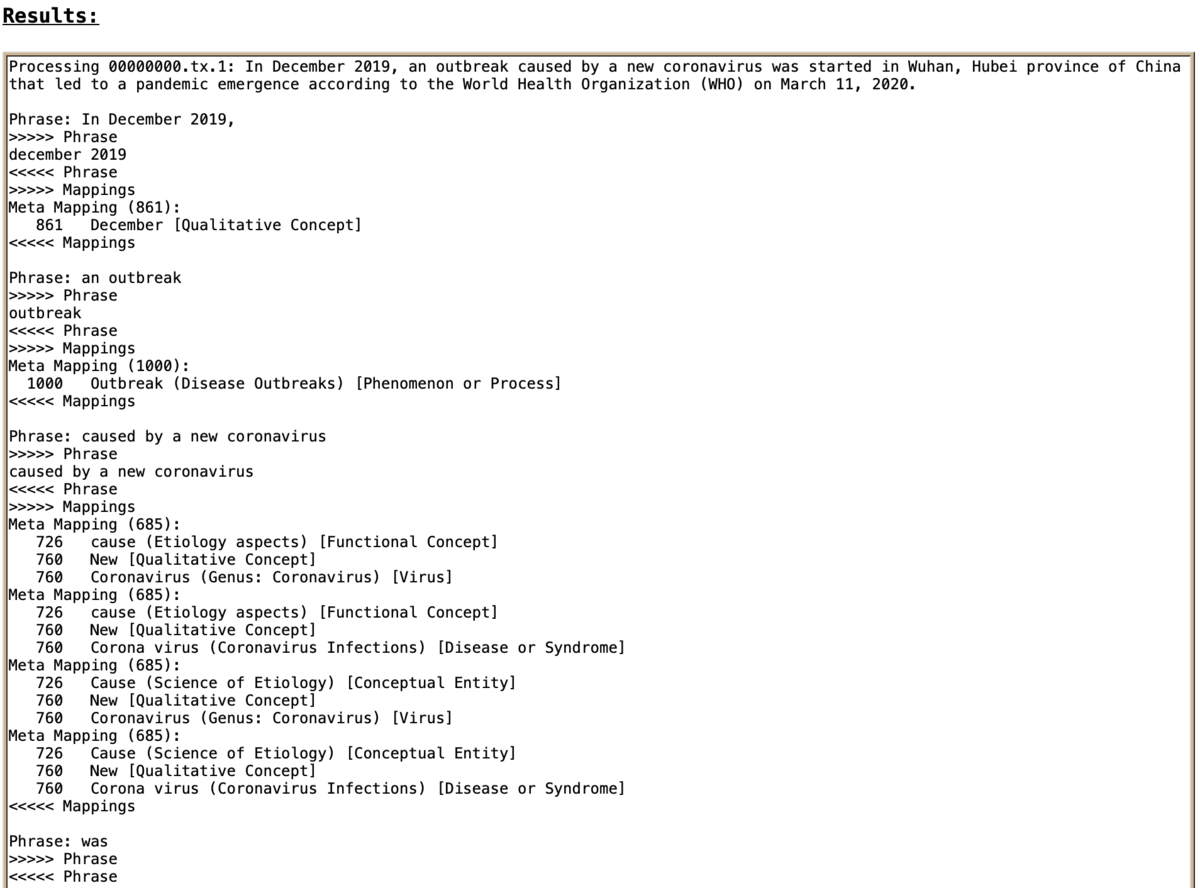
出力結果はおおむね句ごとに区切られて以下のように表示されています.
Phrase: もとのテキストの句 >>>>> Phrase UMLS conceptが検出された箇所 <<<<< Phrase >>>>> Mappings UMLS conceptの検出結果の候補1 (Mapping score): Mapping score 正規化後の表現 [Semantic Type] Mapping score 正規化後の表現 [Semantic Type] ... UMLS conceptの検出結果の候補2 (Mapping score): ... <<<<< Mappings
3-2. 表示オプション(1)
以下の欄から出力結果の表示形式を変更することもできます.
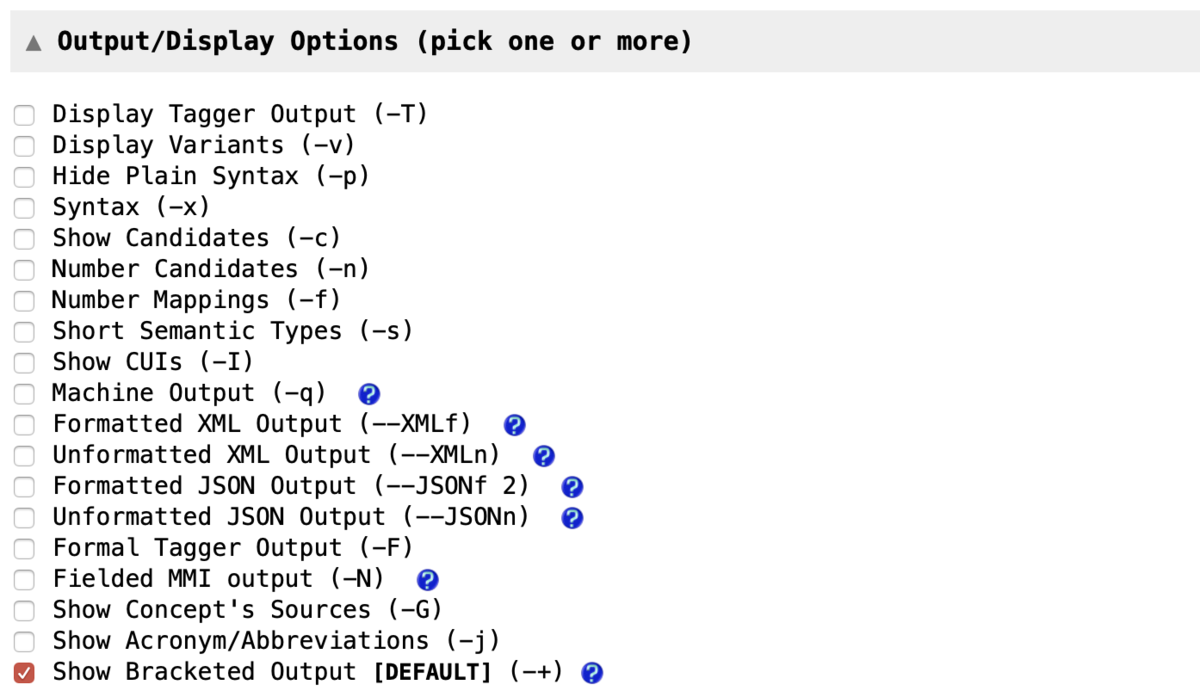
Show CUIs (-I)- デフォルトの表示形式から装飾 (">>>>>", "<<<<<") を省略する.
Display Tagger Output (-T)Show CUIsに加えて品詞タグ付け情報を表示(1行目に文, 2行目に品詞タグ).
Formal Tagger Output (-F)Show CUIsに加えて品詞タグ付け情報を表示(配列形式).
Syntax (-x)Show CUIsに加えて品詞タグ付け+表出形+正規化表現+分かち書きを表示.
Display Variants (-v)Show CUIsに加えて表記ゆれ情報を表示.
Hide Plain Syntax (-p)Show CUIsからPhraseを省略してMeta mappingだけを表示.
Show Candidates (-c)Show CUIsに加えてCandidatesを表示.
Number Candidates (-n)- (未検証)
Number Mappings (-f)Show CUIsに加えてMeta mappingに連番を振る.
Short Semantic Types (-s)Show CUIsのsemantic typeを略語で表示する.
Show Concept's Sources (-G)Show CUIsの各UMLS conceptに対してそれを含むシソーラス名を表示する.
Show Acronym/Abbreviations (-j)Show CUIsの出力の冒頭に検出された略語一覧を付加する.
3-3. 表示オプション(2): Fielded MetaMap Indexing (MMI) Output (-N)
以下のオプションでより詳細な出力結果を得ることも可能です.


- 1列目: PMID.
- 2列目: "MM"という文字列.
- 3列目: MetaMap Indexing (MMI) score (max 1000.00)
- 4列目: UMLS Concept Preferred Name
- 5列目: UMLS Contept Unique Identifier (CUI)
- 6列目: Semantic Type List
- 7列目: Location: TI, AB, or TI;AB
- 8列目: Positional Information: xxx:yyy (position:length)
- 9列目: Treecode
3-4. 表示オプション(3): Machine Output (-q)
以下のような構造をもった MetaMap Machine Output (MMO) 形式での出力.
後述しますが, NIHから提供されているMetaMap実行済みMEDLINE論文データもMMO形式です.
args(コマンド)
aas(略語一覧)
neg_list
utterance('文')
phrase('句',[品詞1(正規化表現,表出形,品詞,分かち書き), 品詞2(正規化表現,表出形,品詞,分かち書き), ...])
candidates
mappings
phrase
candidates
mappings
3-5. 表示オプション(4): XMLで表示
XMLで出力することもできます. 深層学習などに利用するタグ付きコーパスの作成にも便利かと思います.
Formatted XML Output (--XMLf)- 以下のような構造 (一部を抜粋) のXML文書を出力.
Unformatted XML Output (--XMLn)- インデントや改行で整形されていないXML文書を出力.
- 文: <Uttrance> ~ </Uttrance> - 句: <Phrase> ~ </Phrase> - 品詞タグ付け情報: <SyntaxUnits> ~ </SyntaxUnits> - 品詞: <SyntaxType> ~ </SyntaxType> - 正規化表現: <LexMatch> ~ </LecMatch> - 表出形: <InputMatch> ~ </InputMatch> - 品詞: <LexCat> ~ </LexCat> - UMLS concept情報: <Mappings> ~ </Mappings> - score: <CandidateScore> ~ </CandidateScore> - CUI: <CandidateCUI> ~ </CandidateCUI> - 表出形: <MatchedWord> ~ </MatchedWord> - semantic type: <SemType> ~ </SemType> - シソーラス情報: <Sources> ~ </Sources> - シソーラス名: <Source> ~ </Source>
3-6. 表示オプション(5): JSONで表示
JSONでの出力も可能です.
Formatted JSON Output (--JSONf 2)- 以下のような構造 (一部を抜粋) のJSONを出力.
Unformatted JSON Output (--JSONn)- インデントや改行で整形されていないJSONを出力.
"Utterances" : [ { //文 "Phrases" : [ { //句 "Phrasetext" : hogehoge, "SyntaxUnits" : [ { //品詞タグ "SyntaxType" : 品詞, "LexMatch" : 正規化表現 "InputMatch" : 表出形 "LexCat" : 品詞 }, ... ], "Mappings" : [ { //UMLS concept情報 "MappintScore" : score, "MappintCandidates" : { "CandidateCUI" : CUI, "CandidateMatched" : 表出形, "SemTypes" : [semantic type], "Sources" : [収載シソーラス] }, ... ], }, ... ]
3-7. 固有表現抽出のオプション: シソーラスの指定/除外 Restrict to Sources (-R) / Exclude Sources (-e)
デフォルトではUMLSに収載されているあらゆる項目が抽出されますが,
「ICD-10の疾患名だけ」など, 特定のシソーラスの収載項目だけを指定した固有表現抽出も可能です.
3-8. 固有表現抽出のオプション: Semantic Typesの指定/除外 Restrict to Semantic Type(s) (-J) / Exclude Semantic Type(s) (-k)
UMLSの各項目には「疾患名」「解剖学的名称」「薬剤名」などのいずれであるかを表す Semantic Type が付与されています.
www.nlm.nih.gov
これを利用して, 特定の種類の固有表現だけを抽出することも可能です.
- 例1:薬剤名だけ抽出したいとき
- 以下のSemantic Typesだけを含めるようにする:
- Antibiotics (
antb) - Organic Chemical (
orch) - Pharmacologic Substance (
phsu)
- Antibiotics (
- 以下のSemantic Typesだけを含めるようにする:
4. MetaMapの実行(2): コマンドラインで実行する
MetaMapをローカルで実行することもできます.
固有表現抽出を行いたい医療文書が大量にある場合や, コマンドラインでの操作に慣れている場合にお勧めです.
- 実行環境:
- Mac OS X 10.14.6
- Terminal 2.9.5
https://metamap.nlm.nih.gov/MainDownload.shtmlにアクセスし, まずMetaMapの本体をダウンロードしてきましょう.
サイズが大きいので注意してください (tarアーカイブは約1.6GB, 展開後のサイズは約7GBあります).
ダウンロード後は以下のような手順でMetaMapを実行することができます.
# アーカイブを展開 $ tar xf public_mm_darwin_main_2018.tar.bz2 # インストール $ cd public_mm $ ./bin/install.sh # Javaが未インストールならJavaもインストール (以下はHomebrewの場合) $ brew cask install java # SKR/Medpost Part-of-Speech Tagger Serverを起動 $ ./bin/skrmedpostctl start >>> Starting skrmedpostctl: started. # "lung cancer" という文に対して固有表現抽出 $ echo "lung cancer" | ./bin/metamap >>> Phrase: lung cancer Meta Mapping (1000): 1000 C0242379:Lung Cancer (Malignant neoplasm of lung) [Neoplastic Process] Meta Mapping (1000): 1000 C0684249:LUNG CANCER (Carcinoma of lung) [Neoplastic Process] Meta Mapping (1000): 1000 C1306460:Lung cancer (Primary malignant neoplasm of lung) [Neoplastic Process] # 先ほど挙げた各種オプションも適用可能 $ echo "lung cancer" | ./bin/metamap -I -T -p -f -G -s >>> lung cancer noun noun 1. Meta Mapping (1000): 1000 C0242379:Lung Cancer (Malignant neoplasm of lung {CHV,COSTAR,LCH_NW,MEDLINEPLUS,MSH,MTH,NCI,NCI_CTRP,NLMSubSyn,OMIM,SNMI,SNOMEDCT_US}) [neop] 2. Meta Mapping (1000): 1000 C0684249:LUNG CANCER (Carcinoma of lung {AOD,CHV,COSTAR,CSP,CST,HPO,ICD10CM,LNC,MTH,NCI,NCI_CTEP-SDC,NCI_CTRP,NCI_NCI-GLOSS,NLMSubSyn,OMIM,PDQ,SNOMEDCT_US}) [neop] 3. Meta Mapping (1000): 1000 C1306460:Lung cancer (Primary malignant neoplasm of lung {MTH,NLMSubSyn,SNOMEDCT_US}) [neop] # 使い終わったらSKR/Medpost Part-of-Speech Tagger Serverをシャットダウン $ ./bin/skrmedpostctl stop >>> Stopping skrmedpostctl: Stopping Tagger Server process.. Process xxxxx stopped # SKR/Medpost Part-of-Speech Tagger Serverのシャットダウン忘れを確認 # /usr/bin/java ... taggerServer が返るときはシャットダウンできていない $ ps -ef | grep java
5. MetaMapの結果だけが欲しいときは?
MEDLINE論文に対してMetaMapを実行したい場合は, 出力結果が MetaMapped MEDLINE Baselines で提供されているため, そちらを利用するのも手です.
こちらを使うと自力で「MEDLINE論文を取得 -> 本文を抽出 -> MetaMapを実行」を行う手間が省けます.
なお, 出力結果はMachine Output形式で提供されています (上記3-4.を参照).
MIMIC-CXRから読影レポートとjpg画像だけを取得する
はじめに
胸部単純X線の画像と読影レポートの公開データセットMIMIC-CXRを利用するための準備をしてみます。
MIMIC-CXRは次の2つの形式で公開されています:
(1) MIMIC-CXR. DICOM形式の画像と, 読影レポートのtxtファイルが提供されている.
(2) MIMIC-CXR-JPG. JPG形式の画像と, 疾患ラベルが提供されている.
ここで問題となるのは, 「DICOMファイルは欲しくないが読影レポートの原文は欲しい」という場合です。
このためには, (1)からtxtファイルだけ, (2)からJPG画像だけを, それぞれ取得してこなければなりません。
しかし, 特に工夫せずにダウンロードしようとすると, 次のような理由で面倒なことになります:
- MIMIC-CXRとMIMIC-CXR-JPGの構成ファイルすべてをダウンロードしなければならず, 相当な時間がかかる (
wgetコマンドにはワイルドカードや正規表現でファイル名を選別する機能がない) - MIMIC-CXRとMIMIC-CXR-JPGが別のディレクトリツリーを構成するので, ファイル取得後にどちらかに統合する必要がある.
これを解決するためのシェルスクリプトを用意しました.
以下, MIMIC-IIIの利用許可, ID, パスワードはすでに取得済みであるものとします(下記事も参考にしてください).
手順
(1) 以下のシェルスクリプトを download_mimic_cxr_report.sh というファイル名で保存します:
echo -n "physionet.org username? :" read username echo -n "physionet.org password? :" read -s password # MIMIC-CXRからDICOM以外のファイルを取得 wget -r -l 4 -nc -c -np -nv -nH --cut-dir=3 --user $username --password $password https://physionet.org/files/mimic-cxr/2.0.0/
(2) 以下のシェルスクリプトを download_mimic_cxr_jpg.sh というファイル名で保存します:
echo -n "physionet.org username? :" read username echo -n "physionet.org password? :" read -s password gzip -d *.gz unzip *.zip # wgetコマンドの引数として許容される最大長から一度にダウンロードする数を算出 n_div=$((`getconf ARG_MAX`/150 | bc)) # イテレーションの準備 n_sample=`wc -l cxr-record-list.csv | cut -d" " -f1` n_iter=$(($(($((n_sample-1))/n_div | bc))+1)) for ((i=0; i<$n_iter; i++)) do # MIMIC-CXR-JPGからJPG画像のみを取得 line_start=$((i*n_div+1)) line_end=$((i*n_div+n_div)) urls=`cat cxr-record-list.csv | sed -n "${line_start},${line_end}p" | cut -d"," -f4 | cut -d"." -f1 | sed 1d | sed -E "s/(.+)/https:\/\/physionet.org\/files\/mimic-cxr-jpg\/2.0.0\/\1.jpg/g"` wget -r -nc -c -np -nv -nH --cut-dir=3 --user $username --password $password $urls done
(3) download_mimic_cxr_report.shを実行します.
(4) MIMIC-CXRから, 読影レポートがダウンロードされはじめます. DICOMファイルは無視されます.
(5) (4)全体の完了をまたずに, cxr-record-list.gz, mimic-cxr-reports.zip という2つのアーカイブのダウンロードが済み次第 download_mimic_cxr_jpg.shを実行します.
(6) MIMIC-CXRから, JPGファイルがダウンロードされはじめます. その他の重複している余分なファイルは無視されます.
おわりに
これでも丸2日は掛かります。MIMIC-CXRはデータがとにかくでかい!!
MIMIC-IIIを使えるようになるまで
0. MIMIC-IIIとは
MIMIC-IIIとは米国ボストンにある Beth Israel Deaconess Medical Center (BIDMC) が収集した大規模単施設医療データセットです. ICUのあらゆる診療データが12年間にわたって収集されており, その規模は ICU stay 約6.2万回, 成人患者 約3.9万人 に及びます.
↓Nature Scientific Dataの元論文はこちら
データの規模もさることながら, 内容も細かく, 診療記録, 画像診断レポート, 退院サマリーからモニター, ICUチャート, 検体検査結果, オーダー, ICDコードに至るまで収集されています(下図は上記論文からの引用).
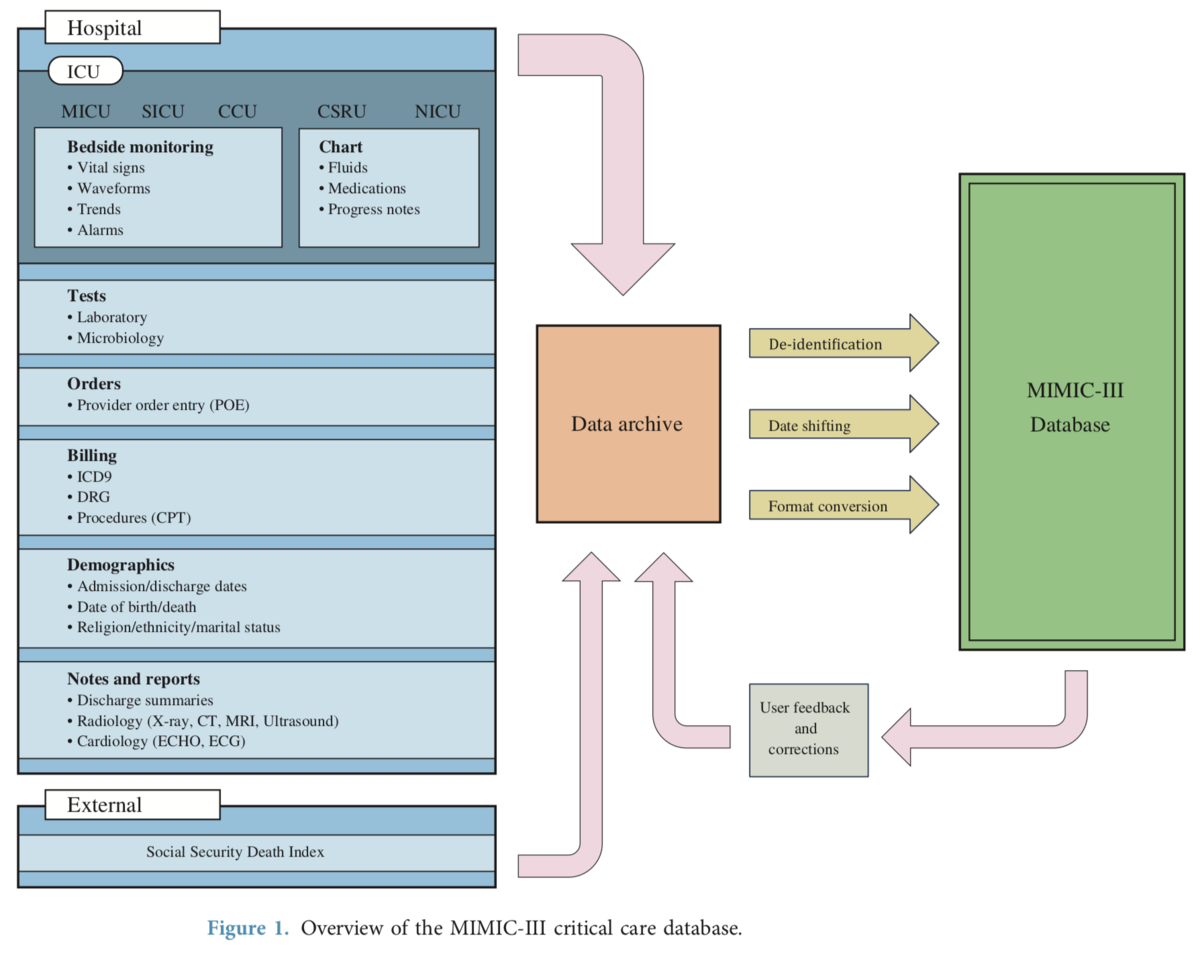
医療言語処理の観点からみても, これだけの規模の診療記録データセットは貴重です. ClinicalBERT も MIMIC-III に収録された診療記録で事前学習を行なっています.
1. MIMIC-IIIのデータを使用するには
MIMIC-IIIは匿名化されたデータであり, HIPAAが定めるところの protected health information (PHI) には該当しないため, 自由に利用可能です.
しかし, かなり詳細な医療データであることには変わりはないため, 利用にあたっては所定の講座を修了のうえ利用申請を行うよう定められています.
利用申請は基本的には案内に従っていけば終了するので, 特段迷うところはありませんが, 以下に手順を記します.
(※2020年1月時点の情報に基づいて記載しています. 現在の正確な情報を反映していない可能性があります)
2. 利用申請の手順
2-1. CITI PROGRAM の Learner Registration
- 下記のリンクからCITI PROGRAM への登録を行います.
page1: Select Your Organization Affiliation
- Massachusetts Institute of Technology Affiliates を選択します.
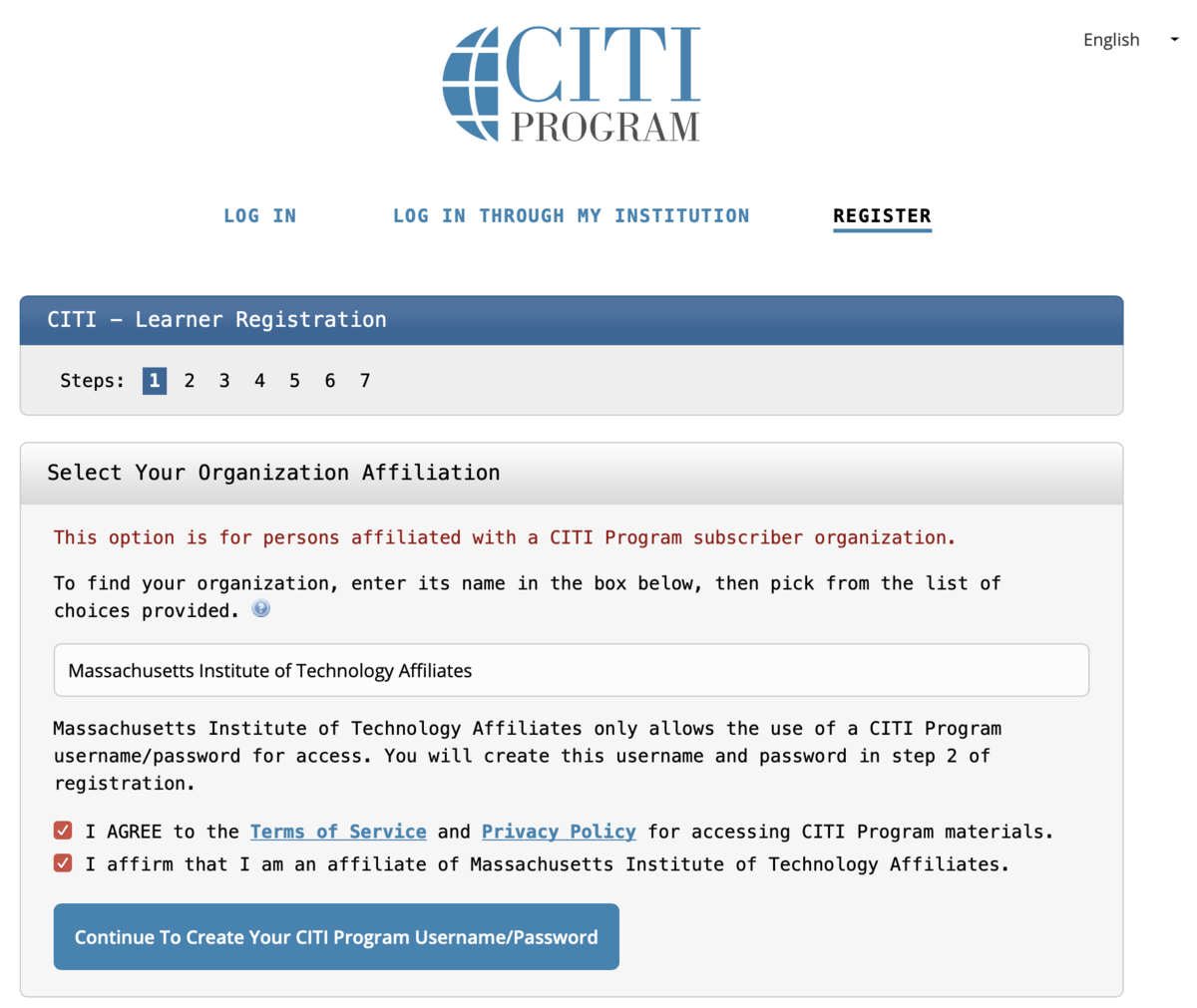
- Massachusetts Institute of Technology Affiliates を選択します.
page2〜4: メールアドレス, ユーザー名, パスワード, 秘密の質問, 国名
- 特に迷うところは無いと思います.
page5: CE creditが要るかどうか
- この講座を医療従事者の生涯学習の単位に利用するかどうか. 基本的に米国の医療従事者でなければ No で構いません.
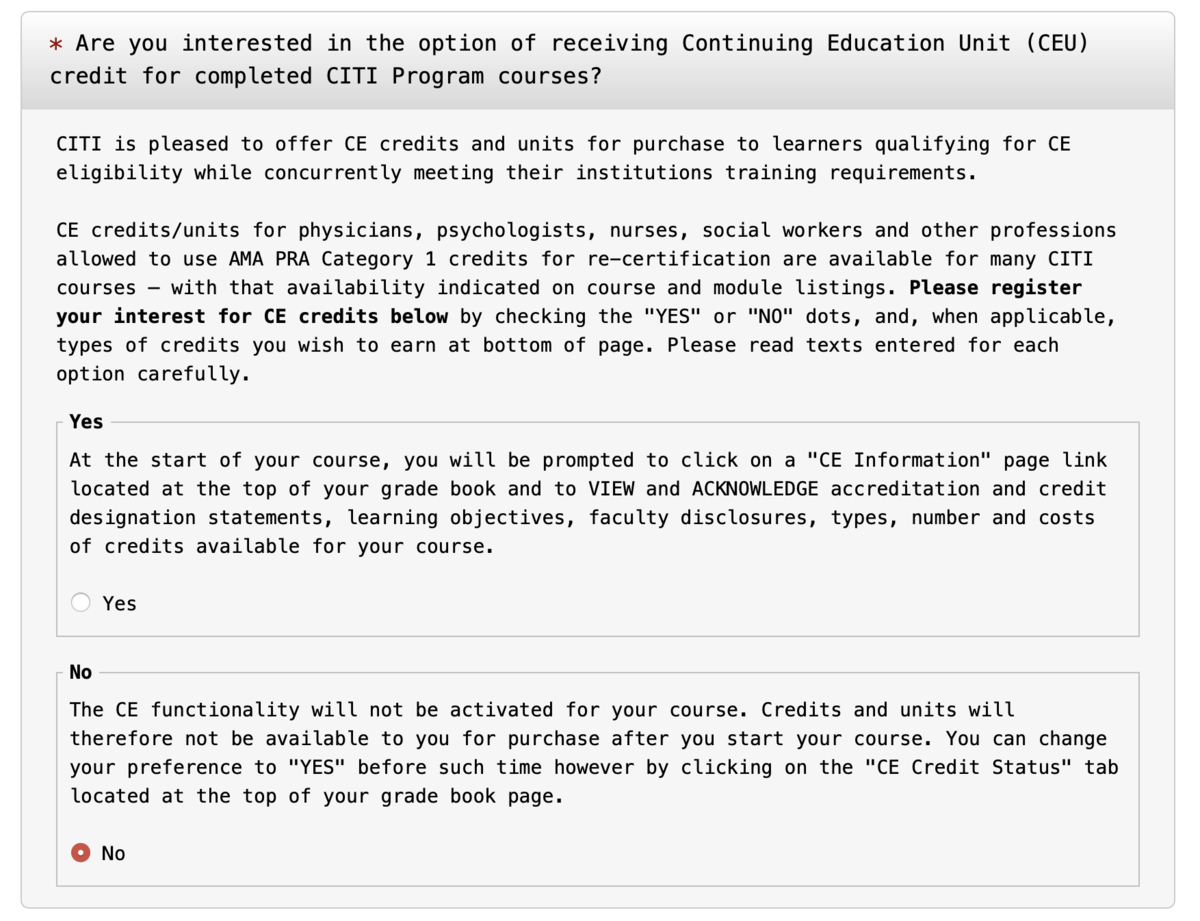
- この講座を医療従事者の生涯学習の単位に利用するかどうか. 基本的に米国の医療従事者でなければ No で構いません.
page6: Language Preference, Institutional Email Address, Department, Role In Research
- 特に迷うところは無いと思います.
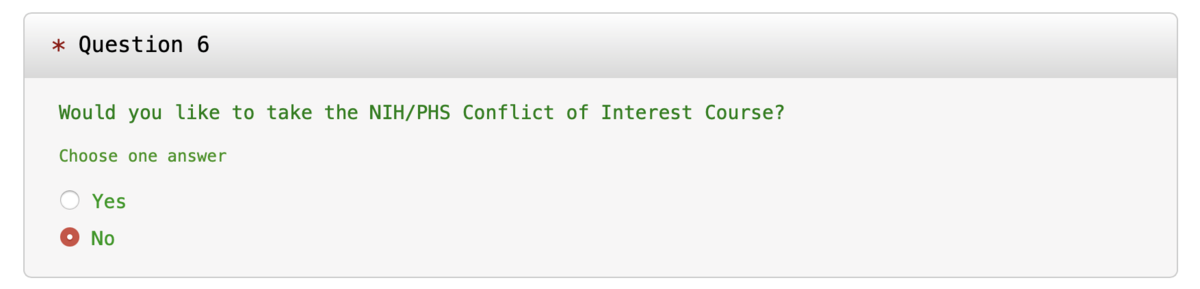
page7: カリキュラム選択
- Question 1 で Data or Specimens Only Research を選択します.
- Question 6 で No を選択します.

Finalize Registration で登録が完了します.
2-2. コースを受講
医療倫理や研究倫理に関する講座を9つ受講し, その後のテストに合格する必要があります.

テストは9講座全体で90%以上の正解率でなければなりません.
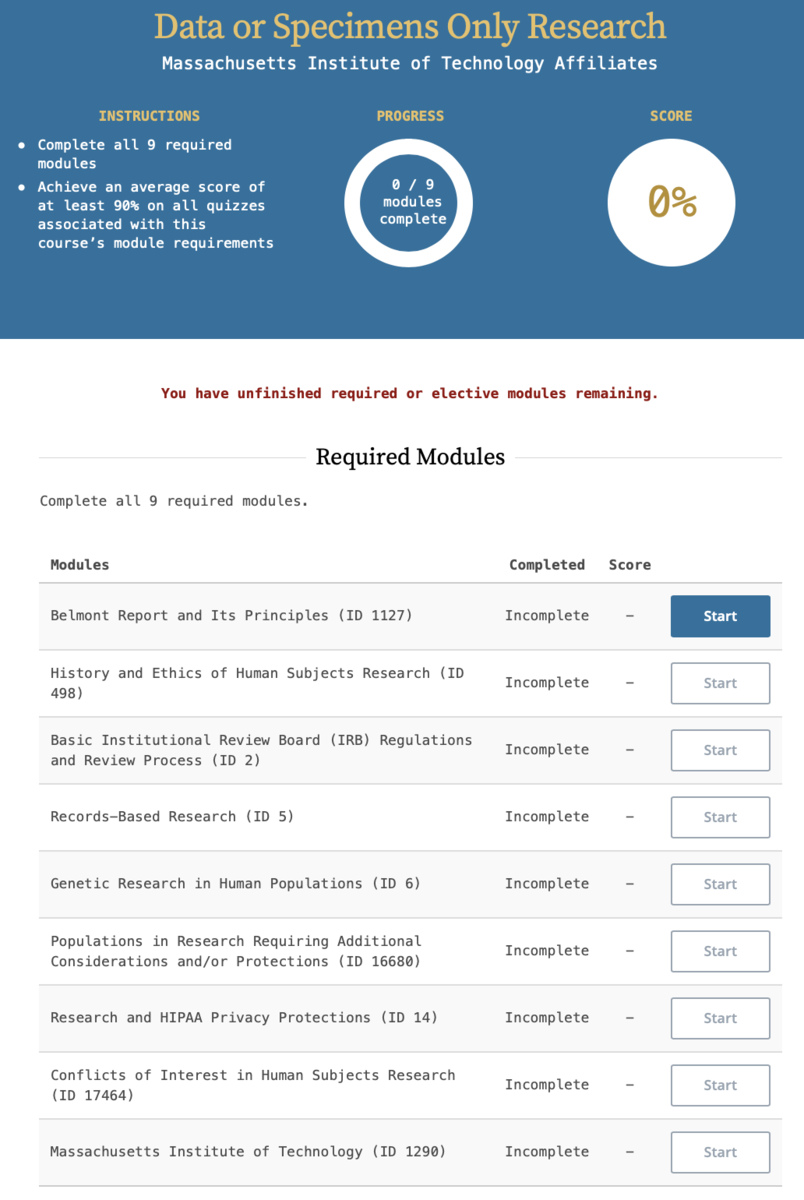
講座の内容はテキスト, テストは多肢選択式問題で与えられます. 結構テキストをしっかり読み込まなければ正解できないため, ここで案外時間がかかります.
2-3. アカウントを証明書付き (Credentialed) にする
- 全9講座の受講が終わったら, メールで下図のような受講終了のお知らせが送られてきます.
- メール内のリンクから Completion Report という PDF を入手しましょう.
- 次に, PhysioNet のアカウント設定画面の Cretentialing タブから, アカウントへの証明書の付与 (Credentialing) を申請します.
- 上司1名の氏名とメールアドレスを添えて申請しなければならないため, 事前にしかるべき人物に話を通しておきましょう.
- 先ほど入手した Completion Report もここでアップロードします.
- 特に問題がなければ1週間程度でメールで申請受理の連絡がきます.

2-4. 個々のデータセットの利用申請
今週のinput (2020/2/1〜2/7)
論文: データセット関連
1. Preparing a collection of radiology examinations for distribution and retrieval. (JAMIA 2015)
内容
- 胸部単純X線のレポートと画像が対になった公開データセット.
- 匿名化は以下のようにして実現:
- さらにレポートへのタグ付与を次の2つの手法で行った.
- manual encoding: MeSHとRadLexのコードを人手で付与
- automatic encoding: MTIを用いた付与
- ベースライン手法による症例検索性能を検討した.
- 実験用の症例検索クエリはImageCLEFのクエリを使用.
- 症例検索の性能は人手で評価した.
ひとこと
- 英語での読影レポート匿名化は実用に耐えうるレベルで実現しているようですね.
2. A Corpus with Multi-Level Annotations of Patients, Interventions and Outcomes to Support Language Processing for Medical Literature. (ACL 2018)
内容
- RCT論文からPICOを抽出するためのデータセット, EBM-NLPを作成.
- コーパスはPubMed上のRCT論文5,000通.
- 分野はcardiovascular, cancer and autism
- アノテーション手順は以下のとおり
- 簡便性のためPICOのIとCは区別せずP,I,Oの3種類でtagging
- ツールにはBRATを使用
- stage 1 annotation:
- P,I,Oのいずれかに該当する区間をすべてアノテート
- stage 2 annotation:
- P,I,Oそれぞれについて以下をアノテート
- アノテーターの認知的負荷を減らすためP, I, Oはそれぞれ別個に行った
- 階層構造をもったタグ
- repetition (情報の重複を検出するため)
- MeSHタグの付与
- アノテーターはクラウドソーシングで募集
- 募集にはAmazon Mechanical Turk (AMT), Up-workを使用
- ベースライン
ひとこと
論文: 医療言語処理タスク関連
3. Introducing Information Extraction to Radiology Information Systems to Improve the Efficacy on Reading Reports. (Methods Inf Med 2019)
https://www.thieme-connect.com/products/ejournals/abstract/10.1055/s-0039-1694992www.thieme-connect.com
内容
- 中国語読影レポートに対して固有表現抽出を行い, さらに検証実験で情報抽出の効率化ができることを実証した.
- 対象は肺CT 3000件.
- 定義した固有表現ラベルは5種類
- Embeddingは部首, 文字, 単語それぞれのレベルの情報を統合した hierarchial embedding
- 漢字 -> radical -> (CNN) -> characted -> (BIES encoder) -> word
- 行列は word2vec (CBOW) で作成
- NERの手法
- 分かち書きはJieba setmentation tool
- multi-embedding-BGRU-CRF
- これとrule-based entity grouping & rankingを組み合わせた
- 検証実験
- 固有表現抽出のF1 score 95.88%, Entity groupingのscore acc 99.23%.
- さらに放射線科医にレポート100件から結節の性状を拾い上げさせる実験を行った
- 所要時間は4〜5割短縮され, accuracyも0.3〜3.8ポイント上昇
- 課題
- Entity groupingをルールベース→ニューラルなrelation extractionに変えると性能向上する可能性あり
ひとこと
- きちんと固有表現抽出による時間短縮効果の検証までしているのはさすが.
- 「漢字の部首の embedding を作った」とさらっと書いていますが結構すごい技術なのではないでしょうか. 中国語のNLPでは一般的なんでしょうか?
論文: その他
4. What does BERT look at? An Analysis of BERT's Attention. (ACL 2019)
内容
- BERTのattentionの機能の分析を試みた論文.
- 類似テーマの論文は多数あるが, 比較的早期に出たものの1つ.
ひとこと
- この論文以外にもBERTのattentionについては議論が多いので, きちんと予測根拠を示すのであれば勾配ベースで分析すべきかもしれません.
その他
手動botのこと
- 読んだ論文を淡々と流すだけのTwitterアカウントを開設しました.
- 基本的に毎日昼の更新を目指しています. ここ約2週間は1日を除いて毎日更新できています. データセットの論文が多めになってしまっていますが.
英語論文執筆のこと
- 3週間ほど前に言語処理学会への演題登録が終わったので, 英語論文を書いています.
- 1月末には投稿する気でいましたが, ちょっと見通しが甘かったですね. 予想外の進まなさに慄いています.
関連文献の読み込みのレベルを上げる作業に時間がかかったり, self-containedであることを目指すために記述量が増えたりしています. いくら遅くても2月末には投稿にこぎつけたいです.
気になっていたアカデミックライティングの日本語訳が出たので買いました. 第1章からとても中身が濃く, 英文の書き方がまるで変わります. これまで書きかけていた論文も丸ごとリライトしました. www.amazon.co.jp
研究コードのパッケージ化
- ずっとまともに動くsetup.pyが書けずにいましたが, ようやく自分のコードをパッケージ化できるようになりました(嬉しい!!)
- 参考にしたリポジトリは↓です. ディレクトリ構造を丸ごとコピーして, サンプルのsetup.pyを少し書き換えるときちんと動くようになります. 時間が余ったときに記事でもまとめようかと思います. github.com
日本語BERTモデルをPyTorch用に変換してfine-tuningする with torchtext & pytorch-lightning
TL;DR
①TensorFlow版訓練済みモデルをPyTorch用に変換した
(→方法だけ読みたい方はこちら)
②①をスムーズに使うための torchtext.data.Dataset を設計した
③PyTorch-Lightningを使ってコードを短くした
はじめに
日本語Wikipediaで事前学習されたBERTモデルとしては, 以下の2つが有名であり, 広く普及しています:
- SentencePieceベースのモデル (Yohei Kikuta さん提供)
- TensorFlow版
- Juman++ベースのモデル (京大黒橋研提供)
- TensorFlow版
- PyTorch版(Hugging Face transformers準拠)
このうち, SentencePieceベースのものは現在TensorFlow版のみの提供となっており, PyTorch版は存在しません。
そのため, 私のようなPyTorchユーザーでがっくり肩を落とされた方は多いのではないでしょうか?
しかし決して諦めることはありません。
実は, ほんの少し工夫するだけでPyTorch版に変換することは可能です!
早速試していきましょう。
(本記事の手法を試すにあたり, kaggler-ja slackの皆さんには多くの助言をいただきました。この場を借りてお礼申し上げます)
環境
- Google Colaboratory
- Python 3.6.9
- TensorFlow 1.15.0
- PyTorch 1.3.1
- Torchtext 0.3.1
- PyTorch-Lightning 0.5.3.2
実践
0. 下準備
0-1. Yohei Kikutaさん版日本語BERTモデルの取得
 こちらに公開されているファイルを取得しておきます。
こちらに公開されているファイルを取得しておきます。
- BERTモデルのCheckpoint
model.ckpt-1400000.indexmodel.ckpt-1400000.metamodel.ckpt-1400000.data-00000-of-00001
- BERTモデルのメタグラフ形式 (今回は使用しません)
graph.pbtxt
- SentencePieceモデル
wiki-ja.modelwiki-ja.vocab
ここではGoogle Driveの My Drive/NLP/bert_yoheikikutasan/ 直下に保存するものとします。

0-2. Google Driveのマウント
つづいてGoogle Colaboratoryに入り, 仮想マシンにGoogle Driveをマウントします。
from google.colab import drive import pathlib # Google Driveをマウントする仮想マシン上のディレクトリ DIR_DRIVE = pathlib.Path('./gdrive/') # Google Drive上でのNotebook等の各種ファイルのパス DIR_COLAB = DIR_DRIVE / 'My Drive/Colab Notebooks/' DIR_PROJCET = DIR_COLAB / 'livedoor_classification/' # Google Driveをマウント drive.mount(DIR_DRIVE)
標準出力にしたがってアカウント認証と認証コードの入力を行い, マウントを完了させます。
1. 訓練済みBERTモデルの変換
1-0. 方針

つづいて訓練済みBERTモデルをTensorFlow用からPyTorch用に変換していきましょう。
PyTorch側でモデルの"ガワ"だけ作っておき, そこにTensorFlow用モデルの重み行列の中身を流し込むイメージです。
PyTorch用モデルの"ガワ"はゼロから設計はせず, PyTorch用BERT族の定番ライブラリ(Hugging Face Transformers)を利用します。
1-1. Hugging Face transformersの準備
Hugging Face Transformersをインストールし, モデルの枠組みをつくります。
!pip install transformers
from transformers import BertConfig, BertForPreTraining, BertTokenizer, BertModel # configの用意 (語彙数は30522 -> 32000に修正しておく) bertconfig = BertConfig.from_pretrained('bert-base-uncased') bertconfig.vocab_size = 32000 # BERTモデルの"ガワ"の用意 (全パラメーターはランダムに初期化されている) bertmodelforpretraining = BertForPreTraining(bertconfig)
1-2. TensorFlowモデル -> PyTorchモデルの変換
つづいてTensorFlow版BERTの重み行列を読み込み, PyTorch版モデルに読み込みましょう。
これはHugging Face Transformersのメソッド一発で簡単にできます。
なお, この工程でTensorFlowのcheckpointパスが必要になりますが, TensorFlowの文脈で「checkpointのパス」といった場合は .index, .meta, .data-XXXXX-of-YYYYY などの拡張子を除いた部分を指すことに注意が必要です。
DIR_BERT_KIKUTA = DIR_DRIVE / 'My Drive/NLP/bert_yoheikikutasan/' BASE_CKPT = 'model.ckpt-1400000' # 拡張子は含めない # TensorFlowモデルの重み行列を読み込む (数分程度かかる場合がある) bertmodelforpretraining.load_tf_weights(bertconfig, DIR_BERT_KIKUTA / BASE_CKPT) # BERTの本体部分だけ取り出す bertmodel = bertmodelforpretraining.bert
これで無事にPyTorch版日本語BERTを手に入れることができました!
2. Livedoorニュースコーパスでfine-tuningする
2-0. 方針
ここからは, 手に入れた日本語BERTモデルでLivedoorニュースコーパスに対する文書分類タスクを解いてみます。
まずは torchtext を用いて前処理の準備をしていきましょう。

torchtextは主に前処理とミニバッチの切り出しの省力化に特化したライブラリであり, 以下の(1)〜(4)をより少ないコード量で実現することができます。
- (1) train, test用データを1行1サンプルのtsvに変換し, 同ディレクトリに別ファイルとして保存しておく
- (2) 前処理を定義する (
torchtext.data.Field) - (3) tsvの各カラムに Field を割り当て, 前処理を一括で実行 (
torchtext.data.Dataset) - (4) 訓練時にミニバッチを自動的に取り出す (
torchtext.data.Iterator)
2-1. tsvファイルの作成
詳しくはこちらを参照してください。 radiology-nlp.hatenablog.com
Livedoorニュースコーパスは9種類の記事からなるため, ここではtsvのカラムは左から順に元のテキストファイル名, 記事本文, ラベル9個のone-hot encoding の形式としました。
filename article dokujo_tsushin it-life-hack ... topic-news hogehoge.txt fugafuga 1 0 ... 0
2-2. 訓練済みサブワード分割器の読み込み
つづいて, BERTの事前学習で使用されたのと同じサブワード分割器 (SentencePiece) のモデルを読み込み, 復元します。
まず SentencePiece をインストールしましょう。
!pip install sentencepiece
次に SentencePiece モデルを読み込みます。
import sentencepiece as sp BASE_SPM = 'wiki-ja.model' BASE_VOCAB = 'wiki-ja.vocab' # 一旦空の SentencePiece モデルを作成 spm = sp.SentencePieceProcessor() # 読み込み. 成功すると True が返る spm.Load(DIR_BERT_KIKUTA / BASE_SPM)
2-3. 前処理の定義 (torchtext.data.Field)
前処理を torchtext.data.Field に定義していきましょう。
基本的に1つのFieldは1種類の前処理しか行うことができません。
このため, 2種類以上の前処理を行う場合はそれぞれについてFieldをつくっておく必要があります。
また, クラスラベル等のように "前処理を何も行わない" カラムに対しても, 前処理を何も行わないことを定義したFieldがやはり必要です。
したがって, 文書分類タスクの場合,少なくとも入力文用とクラスラベル用の2種類のFieldが必要になります。
では前処理をどのように定義するかというと, torchtext.data.Field のコンストラクタに callable を渡すことでその callable の内容を前処理として実行させることができます。
radiology-nlp.hatenablog.com
ここでは↑の記事で作っておいた分かち書き用クラスを使います。
これを使うと, どのような分かち書き器に対しても同じコードで分かち書きができるようになります。
MAX_LEN = 256 stp = SentencePieceTextProcessor(spm, MAX_LEN) passage = '吾輩は猫である。' stp.to_wordpieces(passage) # ['▁', '吾', '輩', 'は', '猫', 'である', '。'] stp.to_token_ids(passage) # [9, 20854, 9947, 4167, 0, 18, 10032, 1164, 3899, 29] stp.to_bert_input(passage) # [4, 9, 20854, 9947, 4167, 0, 18, 10032, 1164, 3899, 29, 3, ..., 3, 5]
無事に前処理が定義できたところで, この前処理の機能を搭載したFieldをつくりましょう。
その他に, クラスラベル用に前処理を何も行わないFieldも定義します。
import torch import torch.nn as nn import torch.optim as optim import torchtext # 文を分かち書きしてBERT形式のID列に変換するField field_text = torchtext.data.Field(sequential=True, use_vocab=False, batch_first=True, tokenize=stp.to_bert_input, include_lengths=True) # 何もしないField field_label = torchtext.data.Field(sequential=False, use_vocab=False)
2-4. 前処理の一括実行 (torchtext.data.Dataset)
これで, データセットのtsvファイルに対して前処理を一括で行う準備ができました。
ここまで来れば, データセットに前処理を施して torch.Tensor 形式に変換したものを短いコードで得ることができます。
まずは, データセットのtsvのどのカラムにどの前処理を割り当てたいかを指定しましょう。
import random PATH_TSV = '' # 3-1. で作成したtsvのパス # tsvの各カラムに割り当てる名前とFieldを指定する # (field_name, torchtext.data.Field) のタプルを容れたリスト # torchtext.data.Fieldは反復使用してよい # field_nameは反復使用不可 N_CLASS = 9 fields_livedoor = [('filename', None), ('text', field_text)] + [('label_{}'.format(i), field_label) for i in range(N_CLASS)]
続いてtorchtext.data.Datasetのコンストラクタを実行しましょう。
すると, ここでtsvファイルからデータが読み出され, 前処理が実行され, その結果がtorchtext.data.Datasetオブジェクトに格納されます。
# tsvファイルの各カラムに対応するFieldに割り当てられた前処理が実行され, 結果がtorch.Tensorで格納される ds = torchtext.data.TabularDataset(path=PATH_TSV, format='tsv', skip_header=True, fields=fields_livedoor) # train/val/testを分離 ds_train, ds_val, ds_test = ds.split(split_ratio=[0.8, 0.1, 0.1], random_state=random.seed(42))
2-5. torchtext.data.Iteratorの作成
次に, ミニバッチの切り出しを楽にしてくれる torchtext.data.Iterator をつくりましょう。
これも短いコードで書くことができます。
BATCH_SIZE = 32 dl_train = torchtext.data.Iterator(ds_train, batch_size=BATCH_SIZE, train=True) dl_val = torchtext.data.Iterator(ds_val, batch_size=BATCH_SIZE, train=False, sort=False) dl_test = torchtext.data.Iterator(ds_test, batch_size=BATCH_SIZE, train=False, sort=False)
イテレーターを回すとミニバッチを取り出すことができます。
ミニバッチはtorchtext.data.Batchオブジェクトで与えられており, ミニバッチの行列はこのオブジェクトのプロパティとして格納されています。
# 試しにdl_trainを回してみる for batch in dl_train: # プロパティ名はtorchtext.data.Dataset定義時に与えたfield_nameと同じ print(batch.text) # torch.LongTensor of size 32x256 print(batch.label_0) # torch.LongTensor of size 32 print(batch.label_1) # torch.LongTensor of size 32 print(batch.label_2) # torch.LongTensor of size 32 print(batch.label_3) # torch.LongTensor of size 32 print(batch.label_4) # torch.LongTensor of size 32 print(batch.label_5) # torch.LongTensor of size 32 print(batch.label_6) # torch.LongTensor of size 32 print(batch.label_7) # torch.LongTensor of size 32 print(batch.label_8) # torch.LongTensor of size 32
3. 学習する
3-1. BERTのfine-tuningするパラメーターを指定
続いてBERTのどの層のパラメータを固定し, どの層をfine-tuningするかを指定します。
ここではBERT Encoder layer 12層すべてと, それに続くPoolerをfine-tuningすることにしましょう。
# 一旦BERTの全レイヤーのfine-tuningを無効にする for _, param in bertmodel.named_parameters(): param.requires_grad = False # Encoder layerのfine-tuningを有効化 for layer in bertmodel.encoder.layer: for _, param in layer.named_parameters(): param.requires_grad = True # Poolerのfine-tuningを有効化 for _, param in bertmodel.pooler.named_parameters(): param.requires_grad = True
3-2. 学習用コード
ここまで来れば, あとは実際の学習のためのコードを書くだけです! あと一歩!
...と言いたいところですが, PyTorchではこの残りの一歩のためにかなり長いコードを書かなくてはいけません。
(実際に書いたことのある方はお分かりかと思います)
そこで, ここでは学習周りのコードを大幅に簡略化できるラッパーの1つ, PyTorch-Lightningを使っていきましょう。
PyTorch-Lightningとは何者で, 何が嬉しいのかは↓の記事に詳しいです。
qiita.com
まずLightningModuleを継承したクラスに, ネットワーク構造, 与えるデータ, 学習のプロセスを指定していきましょう。
生のPyTorchで書くと, でかいfor文を回したり, train/validation/testで細部を変えたりするのが大変ですが, PyTorch-Lightningはあらかじめ与えられた項目を穴埋めするだけでこれらがすべて完成する仕組みになっています。
import pytorch_lightning as pl from pytorch_lightning import Trainer from pytorch_lightning.callbacks import EarlyStopping class LivedoorClassifier(pl.LightningModule): def __init__(self, bertmodel): # モデルの構造を記述 super().__init__() self.bert_model = bertmodel self.bert_hidden_dim = self.bert_model.config.hidden_size # 768 self.affine = nn.Linear(self.bert_hidden_dim, 9) self.logsoftmax = nn.LogSoftmaxe(dim=1) self.postprocess = nn.Sequential(self.affine, self.logsoftmax) self.lossfunc = nn.NLLLoss(reduction='none') def forward(self, inputs, **kwargs): # モデルの推論を記述 # model_output: size (n_batch, 9) model_output = self.postprocess(self.bert_model(inputs)[1]) return model_output def training_step(self, batch, batch_nb): # trainのミニバッチに対して行う処理 """ (batch) -> (dict or OrderedDict) # Caution: key for loss function must exactly be 'loss'. """ # X: size (n_batch, max_len) X = batch.text[0] # T: size (n_batch, 9) (一旦one-hot vector化する) T = torch.cat([getattr(batch, f'label_{i}').unsqueeze(0) for i in range(9)], dim=0).transpose(0,1) # T: size (n_batch) (正解クラスの番号のみ保持) T = torch.argmax(T, dim=1) # GPU使用中ならX, Tを CPU -> GPU に移動させる X = X.to(self.bert_model.state_dict()['embeddings.word_embeddings.weight'].device) T = T.to(self.bert_model.state_dict()['embeddings.word_embeddings.weight'].device).long() # 各クラスに対する対数尤度: size (n_batch, 9) logPY = self.forward(X) # 損失関数: size (n_batch) loss = self.lossfunc(logPY, T) # 推測したクラス: size (n_batch) Y = torch.argmax(logPY, dim=1).long().detach() progress_bar = {'loss':loss} log = {'loss':loss} returns = {'loss':loss, 'pred':Y, 'label':T, 'progress_bar':progress_bar, 'log':log} return returns def validation_step(self, batch, batch_nb, *dataloader_ix): # validationのミニバッチに対して行う処理 """ (batch) -> (dict or OrderedDict) """ # X: size (n_batch, max_len) X = batch.text[0] # T: size (n_batch, 9) (一旦one-hot vector化する) T = torch.cat([getattr(batch, f'label_{i}').unsqueeze(0) for i in range(9)], dim=0).transpose(0,1) # T: size (n_batch) (正解クラスの番号のみ保持) T = torch.argmax(T, dim=1) # GPU使用中ならX, Tを CPU -> GPU に移動させる X = X.to(self.bert_model.state_dict()['embeddings.word_embeddings.weight'].device) T = T.to(self.bert_model.state_dict()['embeddings.word_embeddings.weight'].device).long() # 各クラスに対する対数尤度: size (n_batch, 9) logPY = self.forward(X) # 損失関数: size (n_batch) loss = self.lossfunc(logPY, T) # 推測したクラス: size (n_batch) Y = torch.argmax(logPY, dim=1).long().detach() progress_bar = {'loss':loss} log = {'loss':loss} returns = {'loss':loss, 'pred':Y, 'label':T, 'progress_bar':progress_bar, 'log':log} return returns def test_step(self, batch, batch_nb, *dataloader_ix): # testのミニバッチに対して行う処理 """ (batch) -> (dict or OrderedDict) """ # X: size (n_batch, max_len) X = batch.text[0] # T: size (n_batch, 9) (一旦one-hot vector化する) T = torch.cat([getattr(batch, f'label_{i}').unsqueeze(0) for i in range(9)], dim=0).transpose(0,1) # T: size (n_batch) (正解クラスの番号のみ保持) T = torch.argmax(T, dim=1) # GPU使用中ならX, Tを CPU -> GPU に移動させる X = X.to(self.bert_model.state_dict()['embeddings.word_embeddings.weight'].device) T = T.to(self.bert_model.state_dict()['embeddings.word_embeddings.weight'].device).long() # 各クラスに対する対数尤度: size (n_batch, 9) logPY = self.forward(X) # 損失関数: size (n_batch) loss = self.lossfunc(logPY, T) # 推測したクラス: size (n_batch) Y = torch.argmax(logPY, dim=1).long().detach() progress_bar = {'loss':loss} log = {'loss':loss} returns = {'loss':loss, 'pred':Y, 'label':T, 'progress_bar':progress_bar, 'log':log} return returns def training_end(self, outputs): # trainのミニバッチ1個が終わったときの結果に対する処理 """ outputs(dict) -> loss(dict or OrderedDict) # Caution: key must exactly be 'loss'. """ loss = torch.mean(outputs['loss']) progress_bar = {'loss':loss} log = {'loss':loss} returns = {'loss':loss, 'progress_bar':progress_bar, 'log':log} return returns def validation_end(self, outputs): # validationのミニバッチ全部が終わったときの結果に対する処理 """ For single dataloader: outputs(list of dict) -> (dict or OrderedDict) For multiple dataloaders: outputs(list of (list of dict)) -> (dict or OrderedDict) """ # 全データに対する損失関数 loss = torch.mean(torch.cat([output['loss'] for output in outputs])) # 全データに対する精度 acc = torch.mean(torch.cat([(output['label'] == output['pred']) * 1.0 for output in outputs])) progress_bar = {'val_loss':loss, 'val_acc':acc} log = {'val_loss':loss, 'val_acc':acc} returns = {'val_loss':loss, 'progress_bar':progress_bar, 'log':log} return returns def test_end(self, outputs): # testのミニバッチ全部が終わったときの結果に対する処理 """ For single dataloader: outputs(list of dict) -> (dict or OrderedDict) For multiple dataloaders: outputs(list of (list of dict)) -> (dict or OrderedDict) """ # 全データに対する損失関数 loss = torch.mean(torch.cat([output['loss'] for output in outputs])) # 全データに対する精度 acc = torch.mean(torch.cat([(output['label'] == output['pred']) * 1.0 for output in outputs])) progress_bar = {'test_loss':loss, 'test_acc':acc} log = {'test_loss':loss, 'test_acc':acc} returns = {'test_loss':loss, 'progress_bar':progress_bar, 'log':log} return returns def configure_optimizers(self): # Optimizer, schedulerを指定する # ここでは学習率2e-5でスタートし, 3, 5epoch目でそれぞれ学習率を0.1倍する optimizer = optim.Adam(self.parameters(), lr=2e-5) scheduler = optim.lr_scheduler.MultiStepLR(optimizer, milestones=[3, 5], gamma=0.1) return [optimizer], [scheduler] @pl.data_loader def train_dataloader(self): # torch.utils.data.DataLoader を返させる # torchtext.data.Iterator でも可 return dl_train @pl.data_loader def val_dataloader(self): # torch.utils.data.DataLoader を返させる # torchtext.data.Iterator でも可 return dl_val @pl.data_loader def test_dataloader(self): # torch.utils.data.DataLoader を返させる # torchtext.data.Iterator でも可 return dl_test
これでもコードはかなり長く見えますが, PyTorch-Lightningを使わないと体感的にはこの倍くらいの長さになります!
クラスが定義できたら, コンストラクタでモデルを作成しましょう。
# モデルインスタンスを作成 model = LivedoorClassifier(bertmodel) # モデルをGPUに移す device = torch.device('cuda:0') if torch.cuda.is_available() else 'cpu' model.to(device)
続いて, 「Epoch数」「Early stoppingするかどうか」「ログはどう残すか」など, 学習そのものよりも一歩抽象度の高い, 実験そのものに関するハイパーパラメーターを定義していきましょう。
# Validation lossが3回続けて上昇したら学習をストップさせる early_stop_callback = EarlyStopping(monitor='val_loss', patience=3, mode='min') # ハイパーパラメーターをTrainerに与える trainer = Trainer( early_stop_callback=early_stop_callback, show_progress_bar=True, log_gpu_memory='all', max_nb_epochs=20 )
あとはtrainer.fit()と書くだけで学習がスタートします!
trainer.fit(model)
なお, 初回はtqdmモジュールに関するエラーが出て学習がスタートしない場合がありますが, その場合はランタイムを一旦再起動してコードを実行し直してください。

学習が完了したら, 次に
trainer.test()
と書くことでtest用データでの推論が走ります。
3-3. 結果
Test set での Accuracy は 94.71%となりました.
4. おわりに
本記事で試した内容は以下のとおりです:
- TensorFlow用学習済みモデルをPyTorch用に変換した
- Torchtextを使って前処理のコードを簡略化した
- PyTorch-Lightningを使って学習のコードを簡略化した
だいぶ欲張った内容となりましたが, 少しでも初心者の方の参考になれば幸いです。
